INGENIERÍA GENÉTICA Y SALUD I. ENZIMAS DE
RESTRICCIÓN.

Las enzimas
son proteínas, es decir, cadenas de aminoácidos que llevan a cabo una función
catalizadora. Significa esto que son usadas por los seres vivos para llevar a
cabo transformaciones químicas dentro o fuera de la célula. La evolución hizo
que aparecieran en diferentes especies de bacterias un tipo de enzimas que les
permitirían distinguir entre el ADN propio y el extraño. Todas las enzimas
llevan a cabo un trabajo notable, tanto por su efectividad como por su
especificidad (capacidad para actuar sólo sobre un tipo de reactivo o
sustrato), pero la labor que realizan las enzimas de restricción raya la
perfección. Las bacterias las utilizan como mecanismo defensivo frente a los
virus bacteriófagos, ya que cortan las cadenas de ADN en secuencias de bases
nitrogenadas específicas. De esta forma, los genomas víricos son destruidos
por algunas bacterias, antes de que comience su expresión (es importante
destacar que previamente, estas bacterias protegen su propio ADN metilandolo).
Las enzimas de restricción fueron descubiertas en la década de los 50, al
comprobar que algunas bacterias resistían victoriosamente el ataque de los
fagos, si bien, la importancia del descubrimiento no se valoraría debidamente
hasta los años 70, cuando permitieron el arranque de la ingeniería genética.
Dado que algunas enzimas de restricción son capaces de cortar el ADN en
secuencias concretas, que generalmente comprenden de 4 a 10 pares de bases,
constituyen la herramienta ideal para cortar genes de cara a su posterior
utilización como ADN recombinante.
Las enzimas de restricción se clasifican en tres grupos. Los grupos I y
III no presentan utilidad como herramienta para la ingeniería genética, ya que
realizan cortes en las dos hebras de ADN a distancias variables y no específicas
(entre 25 y 7000 pares de bases respecto a la secuencia de reconocimiento). Sin
embargo, las enzimas de tipo II son herramientas esenciales
para la construcción de ADN recombinante y para el análisis de la
estructura del ADN. Estas enzimas localizan y se unen a secuencias específicas
de ADN, catalizando cortes en las dos hebras dentro mismo o en una zona próxima
a la secuencia de reconocimiento. La longitud de la molécula de ADN es
irrelevante, ya que los cortes que son provocados dan lugar siempre a la aparición
del mismo conjunto de fragmentos.
Se conocen unas 400 enzimas de restricción de tipo II, con un total de
90 secuencias de reconocimiento diferentes. Las enzimas que, siendo distintas,
poseen la misma secuencia diana, reciben el nombre de isoesquizómeros. Cada una
de ellas fue localizada en una bacteria en particular, y su nomenclatura refleja
su origen. De este modo la enzima HaeII y HaeIII, provienen de Haemophilus
aegyptius, MboI y MboII de Moraxella bovis, etc.
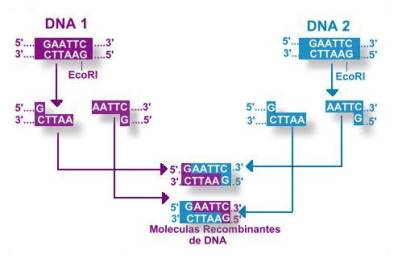
Algunos ejemplos de secuencias que reconocen las enzimas de restricción,
e indicados en rojo, los puntos de corte:
BamHI
5’-GGATCC-3’
EcoRI
5’-GAATTC-3’
3’-CCTAGG-5’
3’-CTTAAG-5’
HindIII
5’-AAGCTT-3’
HpaII
5’-CCGG-3’
3’-TTCGAA-5’
3’-GGCC-5’
XmaI
5’-CCCGGG-3’
TacI
5’-TCGA-3’
3’-GGGCCC-5’
3’-AGCT-5’
PstI
5’-CTGCAG-3’
3’-GACGTC-5’
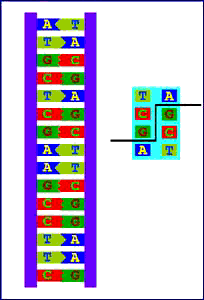
Una enzima de restricción típica
de tipo II: la EcoRI.
La endonucleasa de restricción EcoRI es muy utilizada. Esta enzima reconoce y corta el ADN siempre que localiza la secuencia de bases 5’-GAATTC-3’, una secuencia palíndroma (como es característico de las enzimas del tipo II). Las secuencias de este tipo tienen la propiedad de que leyendo cualquiera de las dos cadenas en el sentido 5’-3’, se lee la misma de bases nitrogenadas. La enzima EcoRI hidroliza los enlaces fosfodiéster que unen las bases G y A de cada hebra, de este modo los cortes que crea son escalonados y dan lugar a la formación de fragmentos de ADN cuyos extremos tienen una corta región monocatenaria que resultará complementaria a las de los otros fragmentos. Los extremos pueden permanecer asociados por puentes de hidrógeno o desnaturalizarse (según sean las condiciones de salinidad, temperatura, etc.), produciendo entonces segmentos independientes, que se reasocian muy fácilmente, por lo que tales extremos reciben el nombre de extremos cohesivos o extremos “pegajosos”.
De este modo, los fragmentos de ADN obtenidos mediante el uso de EcoRI,
podrán ser unidos con toda facilidad, no importando que sean de especies
diferentes (por ejemplo, ADN bacteriano y ADN humano). Tal propiedad permite de
hecho, la obtención de ADN recombinante.
La enzima EcoRI es una proteína formada por dos cadenas polipeptídicas
idénticas, que realizan un corte secuencial del ADN. La enzima interactúa con
un total de 10 pares de bases: seis que constituyen la secuencia diana, y cuatro
de las regiones adyacentes. Parece ser que los pares de bases adyacentes
influyen en la primera hebra que es cortada y sobre la velocidad global del
corte que se realiza en la diana. Esto implica que la velocidad de actuación de
EcoRI no es uniforme en todas las cadenas de ADN.
Existen otros tipos de enzimas de tipo II que reconocen una secuencia de
nucleótidos específica, pero hidrolizan los enlaces fosfodiéster a una cierta
distancia de la diana, y la secuencia de reconocimiento no es palindrómica. El
resultado final es la formación de fragmentos con extremos protuberantes de
cadena sencilla cuya longitud es de un solo nucleótido. Otras, sí reconocen
secuencias palindrómicas específicas en el ADN bicatenario, pero cortan el
enlace fosfodiéster situado en el centro de la diana, con lo que se originan
fragmentos de ADN cuyos extremos están apareados, que reciben el nombre de
extremos romos.