INGENIERÍA
GENÉTICA Y SALUD II. OTRAS ENZIMAS UTILIZADAS PARA LA SÍNTESIS Y DEGRADACIÓN
DE ÁCIDOS NUCLEICOS.
La
capacidad de manipulación del ADN in vitro, es decir, fuera de seres vivos; está
condicionada por la disponibilidad de enzimas que sean capaces de cortar,
modificar y unir moléculas de ADN de manera específica. Hoy por hoy no existe
ningún método químico que permita imitar la selectividad y variedad de tipos
de reestructuración que poseen las reacciones enzimáticas. Un número pequeño
de enzimas (además de las ya comentadas enzimas de restricción) proporciona
las herramientas esenciales para la preparación de ADN recombinante. Muchas de
ellas fueron descubiertas en circunstancias que poco o nada tenían que ver con
sus usos actuales. De hecho, todas las enzimas de las que se hablará tienen un
papel importante en los mecanismos genéticos de los seres vivos. La viabilidad
de las enzimas como herramientas en ingeniería genética depende de su
accesibilidad, de su estabilidad y de su grado de pureza. Es importante también
que no existan otras actividades enzimáticas que puedan interferir en las
reacciones buscadas.

ADN polimerasa.
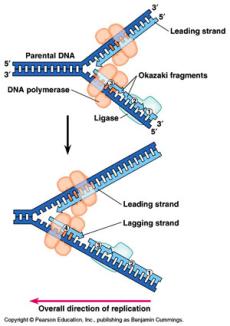
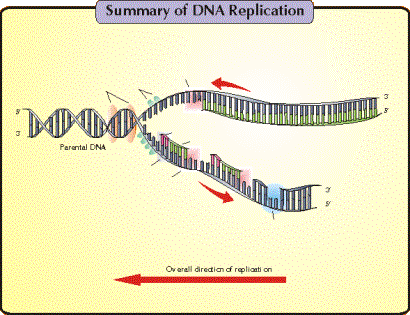
Así pues, las cadenas de ADN se elongan por
adición secuencial de nucleótidos a un cebador con un extremo 3’ libre. Cada
nueva adición supone la elección de un nucleótido dirigida por el molde de
ADN viejo. Todo ello catalizado por la enzima ADN polimerasa. En algunos casos
además, la ADN polimerasa (como la tipo I
de E. coli) presenta actividad exonucleasa, al localizar y eliminar los
nucleótidos de ARN que han actuado como cebador.
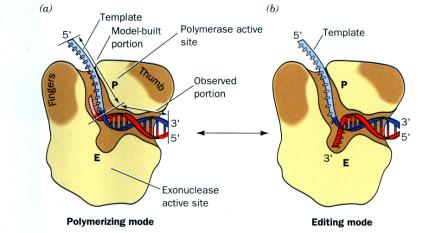
El interés que reviste la ADN polimerasa como herramienta en la ingeniería
genética reside en la capacidad que posee dicha enzima para catalizar
simultaneamente la polimerización y la extracción de determinados nucleótidos
(exonucleosis) en el sentido 5’-3’. La exonucleasa es capaz de degradar la
cadena, comenzando en el extremo 5’de una muesca en una de las hebras de un
ADN bicatenario, mientras que la polimerasa rehace la cadena, añadiendo nuevos
desoxirribonucleótidos al extremo 3’. Por lo tanto, no existe síntesis neta
de ADN, pero la muesca va desplazándose hacia el extremo 3’ a medida que
avanza la el proceso de degradación y sínts catalizado por la enzima (de ahí
el nombre de “desplazamiento de mella”que se da a la reacción).
Además, la ADN polimerasa puede convertir fragmentos de ADN de extremos
cohesivos en fragmentos de extremos “romos”, siempre que los extremos
protuberantes sean los 5’, utilizando el extremo 3’ de cebador y la cadena
5’ protuberante como molde.
En general, las polimerasas permiten sintetizar ADN o ARN in vitro, pero también se usan para marcar los ácidos nucleicos con nucleótidos radiactivos, u otro tipo de trazador. Para ello, se abren muescas en el ADN o ARN con una endonucleasa, y posteriormente se añaden al medio de cultivo los nucleótidos trazados y la polimerasa. Se obtendrá entonces un ADN trazado que podrá servir de sonda para identificar a otros genes.
Transcriptasa
inversa.
La enzima transcriptasa inversa, son enzimas propias de retrovirus. Este tipo de virus poseen ARN en vez de ADN como material genético, por lo que una vez dentro de una célula hospedadora, realizan una transcripción de ARN a ADN (primero monocatenario y luego bicatenario) mediante una enzima especial: la transcriptasa inversa. Dicha enzima puede ser obtenida y purificada a partir de tumores causados por retrovirus, y ser utilizada para catalizar in vitro la síntesis de ADN, tomando como molde una cadena de ARN mensajero. La reacción es similar a la que realizan las ADN polimerasas, y requiere también la presencia de un cebador (suele añadirse para ello el ácido polidT, que se asocia fácilmente con los extremos de los ARN mensajeros eucariotas). El ARN que actúa de molde suele ser una cadena sencilla, por lo que la enzima produce un híbrido ARN-ADN. Posteriormente, las copias creadas pueden ser transformadas en ADN bicatenario mediante la hidrólisis alcalina o el tratamiento de determinadas enzimas de los híbridos. Así, se consigue eliminar el molde original de ARN, y la ADN polimerasa completará la tarea, creando un ADN bicatenario (en este caso la ADN polimerasa, por razones que se desconocen, no precisa de cebador).
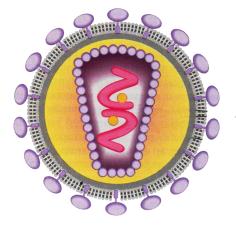
Retrovirus:
fuente de transcriptasa inversa.
El descubrimiento de la ARN polimerasa fue un descubrimiento de vital importancia para los primeros ingenieros genéticos, ya que en los años 70 resultaba del todo imposible aislar y extraer un gen de una célula humana para su posterior introducción en bacterias, de cara a la obtención de productos terapéuticos como la insulina. Pero dado que las células diferenciadas humanas se especializan en la expresión de unos pocos genes, resultó bastante sencillo obtener de las células beta del páncreas abundantes moléculas de ARN mensajero, portando el mensaje genético de la insulina. La transcriptasa inversa permitió obtener el gen de ADN a partir del ARN mensajero, solucionando de paso otro grave problema: la maduración del pre-ARN mensajero, que las bacterias no hubiesen “sabido” hacer, ya que las células eucariotas, eliminan sectores del ARN recién transcrito (intrones) antes de comenzar la síntesis de la proteína, y los seres procariotas no.
Transferasa terminal.
La enzima transferasa terminal es, funcionalmente una polimerasa, ya que cataliza la síntesis de ADN. Al igual que las verdaderas polimerasas requiere la presencia de un cebador con el extremo3’ libre, pero a diferencia de aquellas, no precisa molde alguno. El polímero resultante de la síntesis indica la relación de los desoxirribonucleótidos trifosfato utilizados como reactivos o sustratos. Si se proporcionan dATP, se generará un polímero de nucleótidos de adenina, si se proporciona dGTP, irá creciendo un polímero de nucleótidos de guanina, y así sucesivamente. Su utilidad en ingeniería genética no sólo reside en su capacidad de marcar moléculas al añadirles una cola de nucleótidos trazados, sino que además permite unir por complementariedad dos fragmentos de ADN distintos. Por ejemplo, si a una molécula se le añade una cadena poliA, al otro una cadena poliT y son posteriormente puestos en contacto, inevitablemente se asociarán entre sí. Los huecos que puedan quedar como resultado de los distintos tamaños de las dos colas creadas por la transferasa terminal, pueden ser rellenados con los nucleótidos correspondientes mediante la ADN polimerasa. Finalmente, las “muescas” serán selladas por con la ADN ligasa. Constituye este, pues, un método alternativo al de las enzimas de restricción para unir fragmentos de ADN de procedencia diferente.