
INGENIERÍA
GENÉTICA Y SALUD V. ALGUNAS APLICACIONES DE LAS ENZIMAS DE RESTRICCIÓN.
Se pretende en este punto explicar algunas técnicas usadas en genética molecular que son resultado de la combinación de alguno de los instrumentos antes explicados (enzimas de restricción, electroforesis en gel, ligasas, etc).

Patrones de restricción.
Como se ha dicho anteriormente, las enzimas de restricción son catalizadores aislados en bacterias, capaces de cortar un ADN por secuencias específicas, algunas de las cuales han sido expuestas. Es importante recordar que aunque este tipo de enzimas son propias de seres procariontes, pueden actuar sobre cualquier ADN, independientemente del tipo de célula del que haya sido extraído, y será cortada en tantos puntos como secuencias tipo pueda identificar la enzima concreta. De este modo, una muestra de ADN dado, al ser sometida a la acción de una enzima de restricción concreta, será despedazada en fragmentos de longitud (y masa molecular) concreta. La colección obtenida será distinta a la que se habría obtenido al someter a restricción una muestra de ADN de distinta especie, incluso de distinto individuo (puede compararse pues el patrón de restricción a una autentica huella dactilar genética). Sólo resta entonces, separar los fragmentos por tamaños por el procedimiento de electroforesis en gel (técnica explicada con anterioridad), y obtener un radiografiado de la muestra.

El patrón de restricción de un individuo constituye una inmejorable
mapa físico del material genético de un individuo, y su uso está enfocado a
la comparación con otros. Se puede, por ejemplo, comparar el patrón de
restricción del ADN hallado en el lugar de un crimen con el patrón de un
sospechoso, de cara a su identificación. También se ha usado este principio
para la identificación de parentescos entre personas separadas en la infancia,
o para reagrupar familias de emigrantes, separadas durante mucho tiempo.
Los patrones de restricción también son usados para examinar el grado
de variabilidad genética que existe en el seno de una población. En cualquier
individuo se producen mutaciones por azar. Si las mutaciones afectan a sectores
de ADN con información para construir proteínas (genes), estarán sometidas en
mayor o menor medida a la selección natural, y lo más probable es que no sean
transmitidas a la descendencia. Pero existen amplios sectores de ADN que no son
genes, ni tampoco sirven de molde para la construcción de algún tipo de ARN
(transferente, ribosómico, etc). Es el llamado ADN altamente repetitivo o ADN
basura. Las mutaciones que afecten a estas regiones de los cromosomas, no están
sometidas al filtro de la selección natural, y por tanto son transmitidas por
azar. Como las mutaciones suceden a un ritmo constante, y darían como resultado
patrones de restricción ligeramente diferentes,
pueden compararse estos patrones de restricción de distintos individuos
de una población, y deducirse el grado de variabilidad con que cuenta. Puede
también hallarse el grado de cosanguinidad existente en una población, o la
distancia genética (y evolutiva) que separa a diferentes poblaciones de una
especie o la que separa a diferentes especies, de cara a reconstruir líneas
evolutivas, o clasificaciones taxonómicas de tipo cladístico. La genética
molecular constituye pues, una herramienta complementaria importantísima para
la criminología, la antropología, la zoología, la botánica y la paleontología.
Pero aún hay más.
Dado que las sociedades humanas desarrolladas están escasamente sometidas a la selección natural, y las taras genéticas, de hecho se transmiten, un gen mutante causante de una enfermedad hereditaria, también quedará reflejado en el patrón de restricción. De nuevo, numerosas enfermedades hereditarias podrían ser detectadas en la primera infancia de cara a tratamientos preventivos (o terapia génica cuando esté desarrollada.
Secuenciación de ADN.
Secuenciar un gen significa conocer el orden en que están colocados sus nucleótidos. Existen básicamente tres métodos de secuenciado: el método químico que idearon Maxan y Gilbert en 1977, basado en marcar los extremos del ADN con nucleótidos radiactivos y fragmentarlo con reacciones específicas para cada una de las cuatro bases nitrogenadas (método explicado en este trabajo con anterioridad).
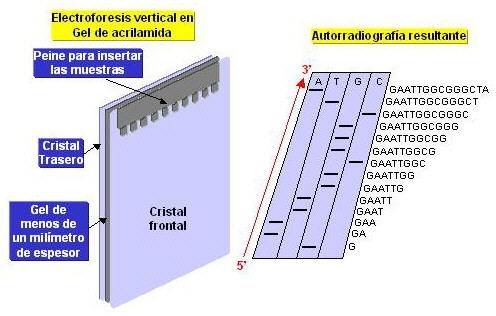
El método enzimático ideado por Sanger en 1981 se basa en la síntesis de una cadena complementaria llevando a cabo cuatro mezclas de reacción independientes que se realizan en paralelo en cuatro viales diferentes. Cada uno de ellos contiene una ADN polimerasa, los cuatro desoxirribonucleótidos trifosfato marcados de algún modo, y en cada uno de los viales un único desoxirribonucleótido diferente que al incorporarse a la cadena en síntesis detendrá la reacción, ya que su extremo 3’ está modificado y no permite proseguir la polimerización. De este modo, en cada uno de los viales se sintetizan cadenas de ADN incompletas que terminarán en cada una de las posiciones en las que existiera el nucleótido correspondiente. Al separar por electroforesis en gel los fragmentos generados en cada uno de los cuatro viales, será posible deducir la secuencia de bases por la longitud creciente de los fragmentos en cada uno de los cuatro carriles de electroforesis. Por último, la secuenciación automática se basa en el mismo principio que el método anterior, pero no es necesario el revelado de la película de electroforesis, ya que un aparato lee automáticamente la posición de los fragmentos por medio de un láser.